서울대-베이징대 공동연구팀, 유전체 희귀변이 연관성 분석 방법 개발

서울대학교 데이터사이언스 대학원 이승근 교수 *재판매 및 DB 금지
[서울=뉴시스]김수연 인턴 기자 = 서울대학교 데이터사이언스 대학원 이승근 교수 연구팀이 중국 베이징대학교 연구팀과 빠르고 정확한 유전체 희귀 변이 연관성 분석 방법을 개발했다고 26일 밝혔다. 본 연구는 유전체 연구 분야 저널 'Nature Genetics'에 게재 승인받았다.
바이오 데이터와 임상정보가 결합된 바이오뱅크는 비약적으로 발전하고 있다. 특히 새로 구축되고 있는 대규모 유전체 시퀀싱 데이터는 질병과 연관된 희귀 유전변이 발견을 가능하게 해 질병 위험도 예측 및 치료법 발견에 기여할 것으로 기대된다.
하지만 이러한 대규모 데이터의 분석 방법, 특히 희귀 유전변이와 질병과의 관계를 테스트하는 방법은 잘 정립돼 있지 않으며, 존재하는 방법은 몇십만 명의 대규모 데이터를 분석하기에는 느리고 부정확한 추론을 하는 문제가 있었다.
이에 연구팀은 새로운 SAIGE-GENE+ 방법을 개발했다. 이전의 방법과 비교해 계산 시간은 최대 1400배, 메모리 사용량은 30배 향상됐으며, 가설검정도 정확하게 수행한다는 설명이다.
연구팀은 SAIGE-GENE+을 영국의 대규모 바이오뱅크인 UK-Biobank의 20만명 전장 엑솜 데이터에 적용해 170여 개의 표현형과 18372개의 유전자를 분석했고, 이를 통해 551개의 유전자 표현형 연관성을 발견했다. 또 유전변이를 빈도와 기능에 따라 여러 개의 그룹으로 나눠서 분석한 뒤 합치는 방식이 검정력이 높다는 사실도 발견했다.
연구팀은 "본 연구는 바이오뱅크의 대규모 시퀀싱 데이터에서 희귀 유전자 분석을 가능케 하는 새로운 방법을 개발했으며 희귀 유전자와 질병의 연관성을 발견하는 데 공헌하게 될 것"이라는 연구 의의를 밝혔다.
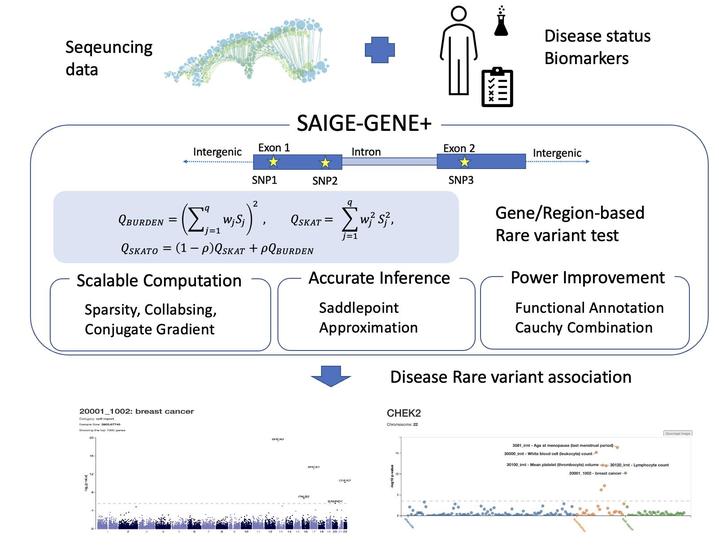
SAIGE-GENE+ 개요 *재판매 및 DB 금지
◎공감언론 뉴시스 [email protected]
Copyright © NEWSIS.COM, 무단 전재 및 재배포 금지