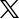코로나19 바이러스 전사체 모두 분석…치료제 개발 탄력
IBS–질본, 사스코로나바이러스-2 고해상도 유전자 지도 완성
숨겨진 RNA·RNA변형 발견, 신약개발 기대…'셀'온라인판 게재
![[대전=뉴시스] 사스코로나바이러스-2(SARS-CoV-2)의 생활사.](https://image.newsis.com/2020/04/10/NISI20200410_0000510076_web.jpg?rnd=20200410084338)
[대전=뉴시스] 사스코로나바이러스-2(SARS-CoV-2)의 생활사.
기초과학연구원(IBS) RNA 연구단 김빛내리 단장·장혜식 연구위원(서울대 생명과학부 교수) 연구팀은 질병관리본부 국립보건연구원과 공동 연구를 통해 신종 코로나바이러스감염증(코로나19)의 원인인 사스코로나바이러스-2(SARS-CoV-2)의 고해상도 유전자 지도를 완성했다고 10일 밝혔다.
공동 연구팀은 2종류의 차세대 염기서열 분석법(나노포어 직접 RNA 시퀀싱·나노볼 DNA 시퀀싱)을 활용해 사스코로나바이러스-2가 숙주세포 내에서 생산되는 RNA전사체를 모두 분석하는데 성공했다.
이번 분석에서 바이러스 유전자의 정확한 위치를 찾아내고 기존 분석법으로는 확인되지 않았던 RNA들을 찾았다. 또 바이러스의 RNA에 화학적 변형이 최소 41곳에서 일어 난다는 것도 발견했다.
이번 연구로 바이러스 전사체가 어떻게 구성됐고 바이러스 유전자들이 유전체 상의 어디에 위치하는지를 정확히 파악할 수 있게 됐으며 연구결과는 생명과학 분야 권위지인 셀(Cell) 온라인판에 지난 9일자로 실렸다.(논문명:The architecture of SARS-CoV-2 transcriptome).
![[대전=뉴시스] 사스코로나바이러스-2(SARS-CoV-2)의 유전체RNA 및 하위유전체RNA 구성, 바이러스 입자 구조의 모식도](https://image.newsis.com/2020/04/10/NISI20200410_0000510077_web.jpg?rnd=20200410084410)
[대전=뉴시스] 사스코로나바이러스-2(SARS-CoV-2)의 유전체RNA 및 하위유전체RNA 구성, 바이러스 입자 구조의 모식도
이 하위유전체는 바이러스 입자구조를 구성하는 여러 단백질을 합성하며 복제된 유전자와 함께 숙주세포 안에서 바이러스 완성체를 이루고 이후 세포를 탈출, 새로운 세포를 감염시킨다. 숙주세포 안에서 생산된 RNA의 총합을 '전사체(Transcriptome)'라 한다.
김 단장 연구팀은 이번 연구에서 유전체RNA로부터 생산되는 하위 유전체RNA를 실험적으로 규명하고 각 전사체의 염기서열(유전정보)을 모두 분석, 유전체RNA 상에 유전자들이 어디에 위치하는지 정확하게 찾아냈다.
이를 통해 기존에 알려진 것처럼 하위유전체 RNA는 10개가 아닌 9개라는 사실과 세포 내에서 생산되는 RNA 수십여 종도 추가로 발견했다. 또 융합, 삭제 등 다양한 형태의 하위유전체 RNA 재조합도 빈번하게 일어난다는 사실을 확인했다.
김빛내리 단장은 "새로 발견한 RNA들이 바이러스 복제와 숙주의 면역 반응을 조절하는 단백질로 작용하는지 확인해볼 필요가 있다. 또 RNA의 화학적 변형은 바이러스 생존 및 면역 반응과 관련이 있을 것으로 보인다"고 말했다.
그는 "이 RNA들과 RNA 변형은 바이러스 치료제를 개발할 때 새롭게 표적으로 삼을 만한 후보군"이라며 "사스코로나바이러스-2 각 전사체의 정량을 정확하게 파악했으며, 이를 토대로 진단용 유전자증폭기술(PCR)을 개선할 수 있을 것"이라고 강조했다.
김 단장은 이어 "국내 최초로 도입한 나노포어 직접 RNA 염기분석법과 DNA 나노볼 염기분석법의 상호 보완법도 확인했다"며 "이번 연구는 사스코로나바이러스-2 유전자에 대한 풍부한 정보와 세밀한 지도를 제시, 바이러스의 증식원리를 이해하고 새로운 치료전략을 개발하는 데 기여할 것"이라고 덧붙였다.
◎공감언론 뉴시스 [email protected]
Copyright © NEWSIS.COM, 무단 전재 및 재배포 금지